赵圣明1,赵岩岩1,马汉军1,别小妹2*
1(河南科技学院 食品学院,河南 新乡,453003)2(南京农业大学 食品科学与技术学院,江苏 南京,210095)
摘 要:微生物是导致食品腐败变质和引起食源性疾病的主要因素,如何抑制微生物的生长是食品及医药领域的一个重要研究方向,因此,对抑菌物质抑菌机制的研究也已成为目前国内外研究的热点问题。利用转录组学技术可以从转录组水平上揭示各种抑菌物质抑制微生物生长的分子机制。文中综述了转录组学主要的研究方法,介绍了目前应用最多的RNA测序技术(RNA sequencing,RNA-seq)的技术特点以及转录组学在抑菌机制研究中的国内外研究现状。随着高通量测序技术的迅速发展,转录组学在抑菌机制研究中将具有更广阔的应用前景。
关键词:微生物;转录组学;RNA-seq技术;抑菌机制
1 转录组学简介
转录组是一个细胞或者组织在特定的发展阶段或生长条件下所有的转录和它们的转录数量的全部集合。对转录组的理解是阐明基因组功能元件所必须的,同时还能够揭示细胞和组织的分子构成以及疾病的发生机制[1]。转录组学研究的关键目标包括以下几个方面:编录转录所有个体,包括信使RNA,非编码RNA和小RNA;就其5,和3,末端而言,可以确定基因的转录结构、剪切模式和其他的转录后修饰;在不同的生长条件下定量每个转录表达水平的变化[2]。目前已经有多种方法被用于推断和定量转录组,主要分为2大类:一类是基于杂交的方法,主要是指基因芯片技术(Microarray);另一类是基于测序的方法,主要包括表达序列标签技术(Expression Sequence Tags Technology,EST)、基因表达系列分析技术(Serial Analysis of Gene Expression,SAGE)、基因表达加冒分析技术(Cap Analysis of Gene Expression,CAGE)、大规模平行测序技术(Massively Parallel Signature Sequencing,MPSS)和RNA测序技术(RNA sequencing,RNA-seq)。
基于杂交的方法主要是将荧光标记的cDNA集成到定制的微矩阵芯片或者商业化高密度寡核苷酸微阵列芯片上。目前已经开发了一些商业化芯片,例如带有跨越外显子结合点探针的芯片已经被应用于检测和定量不同的剪切体[3]。基因组嵌合芯片代表基因组已经被高密度的构建并且允许绘制高分辨率的可转录区,从几个碱基对到100个碱基对。基于杂交的方法具有高通量且费用相对较低的优点,但是用于研究大基因组的高分辨率嵌合芯片除外[4]。然而,这些方法也有一些限制性缺点,例如,必须依赖已知的基因组序列,由于交叉杂交导致较高的背景值,由于背景和信号饱和导致检测的动态范围有一定的限制[5]。此外,不同试验条件下基因表达水平的比较通常很难实现且需要非常复杂的规范化方法。
与基于杂交的芯片方法相比,基于测序的方法能够直接确定cDNA序列。早期,主要使用Sanger测序和EST数据库,但是这两种方法的缺点是低通量、价格昂贵且一般不能定量[6]。后来基于标签方法的发展解决了这些缺点,包括基因表达系列分析技术(SAGE)、基因表达加冒分析技术(CAGE)和大规模平行测序技术(MPSS)。这些基于标签的测序方法能够高通量快速精确的分析基因的表达水平。然而这些方法大多数都是基于价钱昂贵的Sanger测序方法且不能够绘制特异性短标签的重要部分到参考基因组上;此外,只能分析转录的一部分;还不能够区分基因的不同亚型。这些不利条件限制了传统测序技术在转录组基因功能注释中的应用。
目前,用于绘制和定量转录组的新的高通量DNA测序方法已经发展起来。2008年6月NAGALAKSHM等[7]和WILHEL等[8]分别报道了利用RNA-Seq技术分析酿酒酵母、裂殖酵母转录组,这标志着RNA-Seq技术成功的应用于科学研究中。RNA-Seq技术与传统的转录组学技术相比具有很多的优势,该技术也被认为是真核转录组分析方法的革新。目前该技术已经应用到老鼠、人类细胞、植物、微生物等研究领域。
2 RNA测序技术(RNA sequencing,RNA-Seq)
RNA-Seq技术是目前最新发展的深度测序技术,由于其具有高通量、结果精确、重现性好及成本低等优势,已经成为目前转录组研究中使用最多的方法。通常,RNA-Seq技术试验流程如下[9]:(1)从动植物、微生物组织或细胞中提取总RNA,然后采用oligo(dT)磁珠法或者试剂盒方法从总RNA中分离纯化得到mRNA;(2)将mRNA反转录生成cDNA片段文库,并对末端进行末端补平和磷酸化得到合适的末端;(3)构建好的cDNA文库通过合适的高通量测序平台进行测序。从理论上讲,任何一种高通量测序技术都可以应用到RNA-Seq上,例如Illumina IG[10]、ABI SOLID[11]、Roche 454 Life Science[12]、Hiseq 2000[13]等测序系统都已经被成功的应用,但是还未见Helicos Biosciences tSMS应用到RNA-Seq中的报道,但该技术也是一种非常适合的测序方法。根据测序结果获得的reads或者与参考基因组或者与参考转录本或者与没有基因组序列的从头组装对齐,绘制一个包括每个基因的转录结构和表达水平的基因组转录图谱。
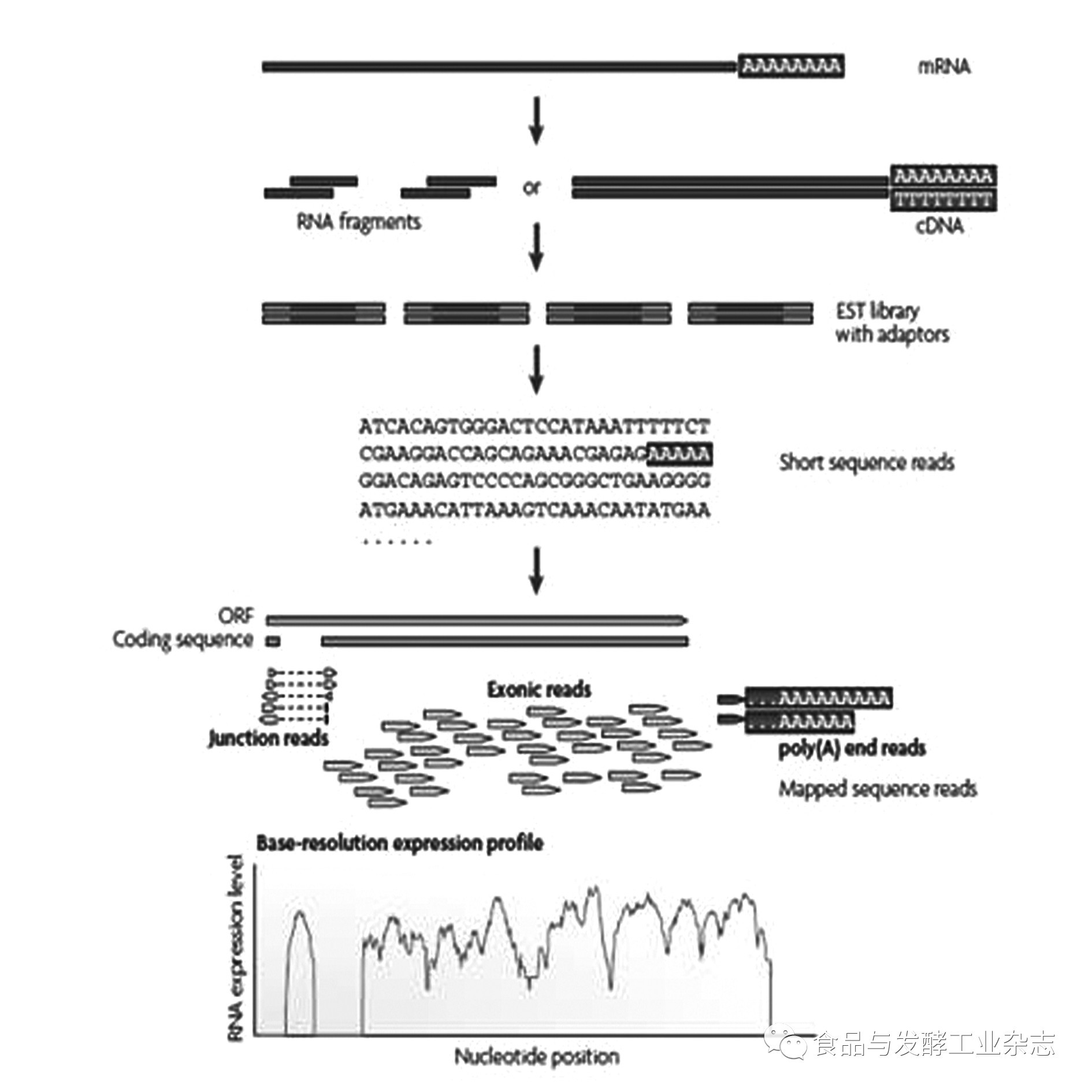
图1 典型的RNA-Seq试验流程图[2]
Fig.1 The typical experimental procedure of RNA-Seq
首先,长链RNAs通过RNA片段化或者DNA片段化被转变成一个cDNA片段文库;测序接头随后被添加到每一个cDNA片段,然后利用高通量测序技术从每一个cDNA获得一个短的序列;最终读取的序列与参考基因组或者转录组比对并被分类为3种类型:exonic reads, junction reads和poly(A)end-reads。这3种类型被用于对每个基因生成一个基本分辨率的表达文件,图1展示了带有内含子的酵母ORF RNA-Seq测序流程。
虽然RNA-Seq还是一个正在发展中的技术,但是与其他技术相比它具有许多优势(表1)。与基于杂交的方法不同,RNA-Seq不仅局限于检测已知基因组序列的转录组,还可以用于全基因组序列未知的非模式生物的转录组测序。例如:基于454的RNA-Seq技术已经被用于格兰维尔贝母蝴蝶的转录组测序研究[12]。此外,RNA-Seq技术能够在单碱基分辨率水平上揭示转录的精确位点,通过30 bp短序列读取能够揭示2个外显子的连接方式,长链序列读取或者双末端短链序列读取能够揭示多个外显子的连通性,同时,RNA-Seq技术还能够揭示转录区的序列变异(如,SNPs)[11]。以上优点使RNA-Seq技术在复杂转录组研究中的应用更加广泛。
RNA-Seq技术与DNA芯片技术相比,RNA-Seq技术可以将DNA测序结果清楚地绘制到基因组的特定区域,因此其显著的优点是背景信号低;同时,RNA-Seq技术定量获得的序列数量没有上限限制,因此,它对于可以检测的转录组表达水平具有一个较大的动态检测范围。在分析一项酿酒酵母绘制出的4千万序列读取数据时发现,其检测范围约是DNA芯片分析结果的9 000倍[7]。与荧光定量PCR(qPCR)[12]和已知浓度spike-in RNA[14]对照的方法相比,RNA-Seq技术在定量分析表达水平方面表现出了更高的精确性。除此之外,RNA-Seq的试验结果还表现出高水平的重现性,包括技术上和生物学上的重复[7, 11];由于检测过程不需要克隆并且使用Helicos技术也无需经过扩增这一步骤,因此RNA-Seq只需要很少的RNA样本就可以完成对样品的检测。
综上所述,RNA-Seq技术是第一个基于测序的方法实现高通量和定量全转录组测序的技术。该技术提供单碱基分辨率的注释和基因组规模的数字化基因表达水平,相对于之前常用的嵌合芯片和Sanger EST测序技术,它的试验成本要相对较低。
表1RNA-Seq技术与其他转录组学技术相比的优点
Table1TheadvantagesofRNA-Seqtechnologycomparedtoothertranometechnologies

3 转录组学在抑菌机制中的应用研究进展
由于转录组技术可以高通量地从分子水平上分析微生物体内基因的变化情况,随着转录组技术的快速发展,该技术的应用越来越成熟,且试验成本也越来越低,因此近年来已逐渐地被应用于抗菌物质抑菌机制研究中。
3.1转录组学在食品加工中常见有害微生物抑菌机制研究中的应用
芽孢杆菌是食品中常见的腐败细菌,由于其可以产生抗逆性强的芽孢,对食品工业的危害较大[15]。有机酸及其盐类是食品中常用的防腐保鲜剂,能显著的抑制细菌、酵母和霉菌的生长,因此研究有机酸对芽孢杆菌的生长抑制能够为有机酸在食品保鲜中的进一步应用提供理论支撑。MAARTEN等[16]应用转录组学技术研究了有机酸对蜡样芽孢杆菌的抑制机理,其转录组分析结果表明经有机酸处理后的蜡样芽孢杆菌氨基酸代谢,脂肪酸代谢以及电子转移链等都发生变化,说明有机酸对菌体的正常生长代谢产生了抑制效果。Beek等[17]研究了山梨酸和醋酸对枯草芽孢杆菌的抑制机制,转录组学研究结果表明弱有机酸主要是通过影响营养代谢、导致细胞质酸化和细胞膜组成发生变化最终破坏细胞膜导致菌体的死亡。
酒类酒球菌是葡萄酒苹果酸乳酸发酵过程中最重要的乳酸菌,但是在酒发酵过程中经常会导致酒类酒球菌的生长受到抑制从而导致苹果酸乳酸发酵过程的失败。有研究报道乙醇是影响该菌在葡萄酒中生长的主要因素,因此揭示乙醇对酒类酒球菌的抑制机制对于葡萄酒工业具有重要意义。Olguín等[18]应用转录组学和蛋白组学技术研究了乙醇对酒类酒球菌的抑制机制,研究结果表明乙醇主要是通过影响代谢物转运、破坏细胞壁以及细胞膜的生物合成等抑制该菌的生长。阪崎克罗诺杆菌是乳粉中主要的致病菌,婴幼儿是克罗诺杆菌感染的高危人群,感染后会引起菌血症、脑膜炎、坏死性小肠结肠炎等,致死率高达40%~80%。冯少龙[19]采用转录组手段研究了大蒜提取物中有机硫化物阿藿烯和二烯丙基硫醚对阪崎克罗诺杆菌的抑制机制,研究结果表明菌体对阿藿烯的反应与对二烯丙基硫醚的反应具有明显的差异,阿藿烯主要导致阪崎克罗诺杆菌运动性相关基因发生下调表达,而二烯丙基硫醚主要引起细胞壁合成相关基因发生上调表达,但是最终都是通过与细胞内含巯基的蛋白或酶结合,导致细胞死亡。金黄色葡萄球菌是引起细菌性食物中毒的重要致病菌之一,而且在水产品、生食蔬菜、生肉、生牛奶、散装熟肉制品等的重要细菌污染监测时发现,金黄色葡萄球菌的污染率最高,因此有效地抑制食品中金黄色葡萄球菌是保证食品安全的一个重要举措。赵星辰[20]通过转录组学方法研究了茶树油对金黄色葡萄球菌的抑制机制,结果表明有715个基因表达发生变化,茶树油主要通过影响菌体的糖酵解途径、嘌呤及嘧啶代谢途径、氨基酸生物合成途径等抑制菌体的生长代谢。副溶血性弧菌是水产食品中重要的食源性致病菌,能够定殖于鱼、虾、蟹和贝类等水产食品的表面,形成生物被膜,从而对食品安全造成威胁。张芳等[21]采用转录组学技术分析天然可食用植物素白藜芦醇对副溶血弧菌的生长及生物被膜形成的影响,研究发现白藜芦醇抑制副溶血弧菌生物被膜的形成是一个多生物过程协同调控的作用,主要是通过干扰副溶血弧菌的新陈代谢过程、群体感应系统、膜蛋白分泌通路等,最终抑制生物被膜的形成。
多聚磷酸盐对大多数革兰氏阳性菌具有较好的抑制作用,被FDA列为食品添加剂用于防止食品腐败。Ji-Hoi等[22]研究了多聚磷酸盐对牙龈卟啉单胞菌的抑制机制,根据转录组分析结果可知,经多聚磷酸盐处理后,与菌体染色体复制、能量代谢以及血红素吸收相关基因的表达都发生明显下调,说明多聚磷酸盐主要通过影响牙龈卟啉单胞菌利用血红素进行生长代谢,最终导致其死亡。目前人工合成具有抗菌肽相似特征的两亲性复合物成为开发新型防腐剂的一个研究热点,例如人工合成的β-氨基酸螺旋复合物和抗菌聚合物等。该类聚合物毒性低、抑菌活性强,在防腐抗菌领域具有广阔的应用前景。因此其抑菌机制的深入研究将为其进一步应用提供理论基础。Bruk等[23]应用转录组学方法研究了聚芳酰胺对大肠杆菌的抑菌机制,结果表明经过聚芳酰胺处理后与细胞膜相关的基因被诱导表达,扫描电镜结果显示菌体细胞膜被破坏,说明聚芳酰胺通过破坏细胞膜来杀死大肠杆菌。这与其他学者的研究结果一致,聚芳酰胺优先结合到细胞膜脂多糖部分,然后破坏细胞膜。
3.2转录组学在食品消毒和包装中常见有害微生物抑菌机制研究中的应用
为防止细菌可能对食品造成的污染,在食品加工领域一般采用化学消毒剂对食品加工设备表面进行消毒处理。蜡样芽孢杆菌能够产生抗逆性强的芽孢,且能够分泌致人呕吐和腹泻的肠毒素,因此在食品工业中对其采取一定的防治措施尤为重要。MARA等[24]研究了常见的消毒剂对蜡样芽孢杆菌转录组的影响,结果表明:经不同消毒剂处理后蜡样芽孢杆菌细胞膜破裂,而与脂肪酸代谢相关的基因被诱导表达,说明菌体尝试合成更多的脂肪酸来修复破裂的细胞膜;另外与DNA损伤修复相关的基因也发生明显的上调,说明经消毒剂处理后菌体DNA受到破坏。氯胺虽然已被广泛的应用于饮用水的消毒,但是关于其抑菌作用方式的研究报道却较少。DIANE等[25]研究了低浓度氯胺对大肠杆菌的抑制机制,转录组分析试验结果表明与菌体DNA修复、生物膜形成、细胞壁修复以及抗生素抗性相关的基因都被诱导表达。这些结果说明低浓度氯胺处理激活了大肠杆菌的防御机制。高梦莎[26]研究了食品工业中常用的杀菌剂臭氧对副溶血弧菌的灭活机制,转录组研究表明在臭氧胁迫下副溶血性弧菌的细胞壁膜、胞内大分子物质以及遗传物质等发生明显变化,细胞膜的通透性受到影响,细胞内活性氧清除酶的活性也受到抑制,进而导致细胞内强氧化产物增加及遗传物质的破坏,最终导致副溶血弧菌死亡。
C60目前被应用于工程纳米材料,由于其具有抗菌性能,因此在包装领域具有广阔的应用前景。研究C60对微生物的抑制机制将为其进一步应用奠定理论基础。DAWN等[27]采用转录组学技术研究了C60对鼠伤寒沙门氏菌的抑制机理,结果表明经C60处理后,与菌体能量代谢、氨基酸合成、DNA代谢等相关的基因发生明显上调,与蛋白质转运、结合等相关的基因表达发生显著下调。这些结果说明C60作用于鼠伤寒沙门氏菌细胞膜导致菌体生长受到抑制可能是因为与关键转运功能蛋白作用的结果。基于TiO2的纳米复合膜具有良好的抗菌性能,因此将其应用于包装材料领域具有较好的市场前景。但是该材料对微生物的抑制机制还不是很清楚,因此研究其抑菌机制有助于更深入的开发其应用价值。ANNA等[28]研究了基于TiO2的纳米复合膜对铜绿假单胞菌的抑制机制,转录组分析发现,经处理后与细菌生长调控、呼吸代谢及细胞壁结构相关的基因发生了明显下调,这些结果表明基于TiO2的纳米复合膜主要通过抑制铜绿假单胞菌的生长代谢最终导致菌体死亡。
3.3转录组学在医学致病菌和农业疫病菌抑菌机制研究中的应用
硝酸银在医学上多用于抗菌治疗,特别是在病原微生物引起的疾病治疗方面被广泛应用。Malli等[29]应用转录组学方法研究硝酸银对蜡样芽孢杆菌的抑制机制,研究结果发现经硝酸银处理后,与菌体营养物质转运、DNA复制及膜蛋白相关的基因都发生上调,说明菌体本身在提高抵御能力;与细菌趋化作用相关的基因发生下调,趋化能力的下降说明菌体通过菌落聚集来抵御外界不良环境的能力减弱。俞文榜[30]研究发现脂环肽抗生素Fusaricidins作用于枯草杆菌时有大量的菌体裂解,但是几小时后枯草芽孢杆菌会重新生长,利用转录组学技术研究了脂环肽抗生素Fusaricidins对枯草杆菌的抑制机理,结果发现枯草芽孢杆菌的铁代谢与抗生素作用机制及其耐药性相关。叶海青[31]通过转录组学的方法研究了白花丹素对结核分枝杆菌的抑制机制,发现经白花丹素处理后,结核分枝杆菌醇二分支菌酸相关基因(DIM)的表达下调,结核分枝杆菌外膜的通透性增加,从而在一定程度上导致该菌活性变低,同时甘油三酯(TG)合成酶基因也受到了抑制,促使结核分支杆菌的应激能力降低,最终结核分枝杆菌的生长受到抑制。朱健铭等[32]应用转录组学手段研究黄连对致病性大肠埃希氏菌抑制作用的分子机制,结果表明有1428个基因发生明显的差异表达,黄连主要通过引起大肠埃希氏菌的细胞壁损伤,影响脂肪酸代谢、氨基酸代谢、蛋白质的转录和翻译、破坏DNA复制过程等多个方面进行作用,最终导致菌体生长被抑制甚至死亡。鼠疫耶尔森菌可以引发鼠疫传染病的发生和流行,而目前该菌对一些常规的抗生素不敏感,因此需要开发新型的天然抗菌剂。白群华等[33]应用转录组学技术研究中药大黄对鼠疫耶尔森菌的分子作用机制。试验结果表明大黄主要通过导致蛋白合成、细胞膜及转运结合相关基因的表达发生变化最终对疫耶尔森菌的生长发挥抑制作用,从分子水平上揭示了中药大黄对疫耶尔森菌主要作用机制。尖孢镰孢菌是农作物和园艺作物生产中危害最严重的一种土壤传播病原物之一,该菌可以引起植物枯萎,从而导致作物的大面积减产。目前仍缺乏有效地防治措施,因此揭示有效杀菌剂对其的抑制机理为开发新型防治技术的提供重要的理论基础。李哲肖[34]研究了万寿菊杀菌剂处理后尖孢镰孢菌转录本中的差异基因表达,结果表明万寿菊杀菌素对尖孢镰孢菌的抑菌作用涉及到多种功能蛋白和代谢通路的多作用位点,尤其是对几丁质酶具有显著地竞争性抑制作用。辣椒疫病是导致辣椒减产和绝收的主要病害,而引起该病的辣椒疫霉极易对传统的农药产生抗药性,因此需要开发新型的杀菌剂防治辣椒疫病。高亮等[35]采用转录组学方法研究了具有广谱抗菌活性的壳聚糖对引起辣椒疫病的辣椒疫霉的抑制机制,结果表明有2322个基因发生了差异性表达,主要是通过影响与细胞膜功能、运动功能及糖代谢途径相关基因的表达而对菌体生长产生抑制作用。目前采用传统的化学农药防止辣椒疫病已经导致了部分辣椒疫霉产生抗药性,因此通过转录组学从分子水平上研究壳聚糖对辣椒疫霉的抑制机制为新型绿色农药的开发应用奠定了理论基础。
4 结论
转录组技术应用于抑菌机制的研究已有数年,新一代高通量测序技术的发展为抑菌机制的转录组学研究提供了新的技术手段,特别是RNA-Seq技术的使用可以通过比较基因组学技术对未进行全基因组测序的微生物进行基因组数据注释,拓宽了微生物的研究领域,有助于对该物种进行后续的分子生物学研究。随着转录组测序技术的逐渐成熟,微生物转录组测序成本在降低,测序结果也越来越准确,可以得到大量的可靠的转录组数据。转录组学方法可以精确地在分子水平上解释抗菌物质对微生物的抑制机理。与此同时,转录组学也有其自身的局限性,如转录丰度与基因的转录产物含量或者其活性之间没有必然的关联,因此通过与蛋白质组学和代谢组学等其他分子生物学技术以及一些表观的抑菌研究结果相结合可以更好地阐明抗菌物质的抑菌机制,为抗菌物质的进一步应用奠定坚实的理论基础。
参考文献:
[1] COSTA V, ANGELINI C, DE F I, et al. Uncovering the complexity of tranomes with RNA-Seq[J]. Journal of Biomedicine and Biotechnology, 2010.DOI:10.1155/2010/853916.
[2] WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for tranomics[J]. Nature Reviews Genetics, 2009, 10(1): 57-63.
[3] CLARK T A, SUGNET C W, ARES M. Genomewide analysis of mRNA processing in yeast using splicing-specific microarrays[J]. Science, 2002, 296(5 569): 907-910.
[4] CHENG J, KAPRANOV P, DRENKOW J, et al. Tranional maps of 10 human chromosomes at 5-nucleotide resolution[J]. Science, 2005, 308(5 725): 1 149-1 154.
[5] ROYCE T E, ROZOWAKY J S, GERSTEIN M B. Toward a universal microarray: prediction of gene expression through nearest-neighbor probe sequence identification[J]. Nucleic Acids Research, 2007, 35(15): 50-54.
[6] GERHARD D S, WAGNER L, FEINGOLD E, et al. The status, quality, and expansion of the NIH full-length cDNA project: the Mammalian Gene Collection (MGC)[J]. Genome Research, 2004, 14(10B): 2 121-2 127.
[7] NAGALAKSHMI U, WANG Z, WAEM K, et al. The tranional landscape of the yeast genome defined by RNA sequencing[J]. Science, 2008, 320(5 881): 1 344-1 349.
[8] WILHELM B T, MARGUERAT S, WATT S, et al. Dynamic repertoire of a eukaryotic tranome surveyed at single-nucleotide resolution[J]. Nature, 2008, 453(7199): 1239-1243.
[9] 祁云霞, 刘永斌, 荣威恒. 转录组研究新技术: RNA-Seq及其应用[J].遗传, 2011, 33(11): 1191-1202.
[10] LISTER R, O'MALLEY R C. TONTI-FILIPPINI J, et al Highly integrated single-base resolution maps of the epigenome inArabidopsis[J]. Cell, 2008, 133(3):523-536.
[11] CLOONAN N, FORREST A R R, KOLLE G, et al. Stem cell tranome profiling via massive-scale mRNA sequencing[J]. Nature Methods, 2008, 5(7): 613-619.
[12] Vera J C, WHEAT C W, FESCEMYER H W, et al. Rapid tranome characterization for a nonmodel organism using 454 pyrosequencing[J]. Molecular Ecology, 2008, 17(7): 1 636-1 647.
[13] 王兴春, 杨致荣, 王敏, 等. 高通量测序技术及其应用[J]. 中国生物工程杂志, 2012, 32(1): 109-114.
[14] MORTAZAVI A, WILLIAMS B A, MCCUE K, et al. Mapping and quantifying mammalian tranomes by RNA-Seq[J]. Nature Methods, 2008, 5(7): 621-628.
[15] OOMES S J C M, ZUIJLEN A C M V, HEHENKAMP J O, et al. The characterisation ofBacillus, spores occurring in the manufacturing of (low acid) canned products[J]. International Journal of Food Microbiology, 2007, 120(1-2):85-94.
[16] MOLS M, VAN KRANENBURG R, TEMPELAARS M H, et al. Comparative analysis of tranional and physiological responses ofBacilluscereusto organic and inorganic acid shocks[J]. International Journal of Food Microbiology, 2010, 137(1): 13-21.
[17] BEEK A T, WILMAN J G E, ZAKRZEWAKA A, et al. Comparative physiological and tranional analysis of weak organic acid stress inBacillussubtilis[J]. Food Microbiology, 2014, 45(Pt A):71-82.
[18] OlGUIN N, CHAMPOMIER-VERGES M, ANGLADE P, et al. Tranomic and proteomic analysis ofOenococcusoeni, PSU-1 response to ethanol shock[J]. Food Microbiology, 2015, 51:87-95.
[19] 冯少龙. 大蒜提取物中有机硫化物对阪崎克罗诺杆菌的抑菌活性与抑菌机理的研究[D].天津:天津科技大学, 2014.
[20] 赵星辰. 茶树油抗金黄色葡萄球菌活性机制研究[D].吉林:吉林大学, 2016.
[21] 张芳, 朱军莉, 冯立芳. 基于RNA-Seq技术分析白藜芦醇对副溶血弧菌生物被膜的抑制作用[J].微生物学报, 2016, 56(5):856-866.
[22] MOON J H, PARK J H, LEE J Y. Antibacterial action of polyphosphate on Porphyromonas gingivalis[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(2): 806-812.
[23] Mensa B, Kim Y H, Choi S, et al. Antibacterial mechanism of action of arylamide foldamers[J]. Antimicrobial Agents and Chemotherapy, 2011, 55(11): 5 043-5 053.
[24] CERAGIOLI M, MOLS M, MOEZELAAR R, et al. Comparative tranomic and phenotypic analysis of the responses ofBacilluscereusto various disinfectant treatments[J]. Applied and Environmental Microbiology, 2010, 76(10): 3 352-3 360.
[25] HOLDER D, BERRY D, DAI D, et al. A dynamic and complex monochloramine stress response inEscherichiacolirevealed by tranome analysis[J]. Water Research, 2013, 47(14): 4 978-4 985.
[26] 高梦莎. 臭氧对副溶血性弧菌灭活机制的探索研究[D].杭州:浙江工商大学, 2013.
[27] HANCOCK D E, INDEST K J, GUST K A, et al. Effects of C60 on theSalmonellatyphimuriumTA100 tranome expression: Insights into C60-mediated growth inhibition and mutagenicity[J]. Environmental Toxicology and Chemistry, 2012, 31(7): 1 438-1 444.
[28] KUBACKA A, DIEZ M S, ROJO D, et al. Understanding the antimicrobial mechanism of TiO2-based nanocomposite films in a pathogenic bacterium[J]. Scientific Reports, 2013, 4(2):4 134-4 134.
[29] BABU M M G, SRIDHAR J, GUNASEKARAN P. Global tranome analysis ofBacilluscereusATCC 14579 in response to silver nitrate stress[J]. Journal of Nanobiotechnology, 2011, 9(1): 1-12.
[30] 俞文榜. 基于转录组的枯草杆菌响应细胞外信号机制研究[D].上海:华东理工大学, 2013.
[31] 叶海青. 抗结核分枝杆菌先导化合物筛选及白花丹素对H37Rv转录组的影响[D].吉林:吉林大学, 2013.
[32] 朱健铭, 翁幸鐾, 吴晋兰,等. 基于 RNA-seq 技术的黄连水煎液对多耐药尿道致病性大肠埃希菌的转录组学研究[J].中华微生物学和免疫学杂志, 2015, 35(10):776-782.
[33] 白群华, 贾燕, 代兴碧,等. 大黄抑制鼠疫耶尔森菌的体外转录组学研究[J].中国人兽共患病学报, 2008, 24(10):905-908.
[34] 李哲肖. 基于转录组测序探究万寿菊杀菌素Ⅱ对尖孢镰孢菌几丁质酶抑制作用[D].太谷:山西农业大学, 2015.
[35] 高亮, 张博, 张悦丽,等. 壳聚糖对辣椒疫霉菌的抑制作用[C].中国植物病理学会2015年学术年会论文集,北京:中国农业出版社,2015:559
Applicationsoftranomicsinresearchofantimicrobialmechanism
ZHAO Sheng-ming1,ZHAO Yan-yan1,MA Han-jun1,BIE Xiao-mei2*
1(School of Food Science, Henan Institute of Science and Technology, Xinxiang 453003, China) 2(College of Food Science and Technology, Nanjing Agricultural University, Nanjing 210095, China)
ABSTRACT:Since microorganism is one of the most primary factors to cause food spoilage and foodborne disease, the inhibition of the growth of microorganism is an important research direction in food and medical field. Therefore, the study on antimicrobial mechanism is a hot topic in domestic and aboard academe. The utilization of tranomics could reveal the molecular mechanism of various antimicrobial substances against microorganism. The main research methods for tranomics, especially the technical features of the most widelyused RNA sequencing technology were reviewed in this paper. The research situations of tranomics for antimicrobial mechanism at home and aboard were introduced. With the development of high-throughput sequencing technology,tranomics will have extensive application prospects in research of antimicrobial mechanism.
Key words:microorganism; tranomics; RNA-sequencing; antimicrobial mechanism
DOI:10.13995/j.cnki.11-1802/ts.013619
第一作者:博士,讲师(别小妹教授为通讯作者,E-mail:bxm43@njau.edu.cn)。
基金项目:河南省重大科技专项项目(161100110600);河南科技学院高层次人才科研活动项目(2016018,2016019);河南省高等学校重点科研项目(18B550003)